製品・サービス
Counting PCR:検量線なしでDNAコピー数を得るための新しい方法
John SantaLucia, Jr. and Gregory J. Boggy
DNA Software, Inc., Ann Arbor MI 48104
はじめに
DNA Softwareはサイクル開始時のDNAコピー数を決定するため、どのようにPCR曲線の形を分析するかについて研究を重ねてきました。この研究の結果、Counting PCR (cPCR)と呼ばれるDNAの絶対定量法を開発しました。
cPCRにおいてはPCRサイクルごとに各DNAコピーが計測され、この結果が絶対的なコピー数となります。結果は機器や蛍光色素に依存しないため、異なる施設で得られた結果の比較や保存されているデータを用いたメタ解析が可能になります。
DNA Software社ではcPCRの原理をクラウドベースで定量的PCRの結果を自動的に解析し、検量線なしでDNA絶対的なコピー数を導出するqPCR CopyCountに採用しています。qPCR CopyCount は10万以上のサンプルに対して使用され、結果がバリデートされています。qPCR CopyCountを用いた高品質な絶対定量はmRNAレベルの遺伝子発現解析、ウイルスの感染、コピー数変異のジェノタイピング、シードの接合性試験、次世代シーケンサー用のフラグメントライブラリ調製および非侵襲的な診断等に利用できます。
qPCR CopyCountはオンラインサービスとしてご利用頂けます (https://portal.dnasoftware.com/login.jsf)

図1: qPCR CopyCount ワークフロー
qPCR CopyCount はクラウドベースのサービスであり、qPCRの結果を自動的に解析し、検量線なしでDNAコピー数の絶対定量が可能です。qPCR CopyCount はBoggy and Woolf によって発表されたMAK2(Mass Action Kinetic model with 2 parameters)と呼ばれるメカニズムベースのフィッティング方法を基礎としています [1]。
この方法で用いる2つのパラメータはサイクル開始時点のDNA濃度D0とサイクルごとの増幅効率の変化を表すkです。MAK2は以前は異なるサンプル中の同一の対象遺伝子の相対的な定量にしか使われていませんでした。DNA Software社はMAK2を改良した、ほとんどのPCR機器で得られたデータに対して完全に自動化されたカーブフィッティングを実現するソフトウェア qPCR CopyCountを開発しました。
このソフトウェアは、徹底した誤差分析により、MAK2法と比較して3倍の精度を実現し、DNA定量の信頼性を各段に向上させました。最も重要なことは、qPCR CopyCount はcPCRについても適用可能であり、あらゆるqPCR実験に際し、検量線なしで絶対定量が可能であるということです。図1ではqPCR CopyCountのシンプルなワークフローが示されています。ユーザーはqPCRデータを収集し、それをクラウドベースのソフトウェアにアップロードするのみです。qPCR CopyCountは次のような内容を含む解析を実行します。
- 解析に採用するデータ範囲の決定
- どのウェルのデータがサンプルからのシグナルであり、どのウェルのデータがノイズであるのかの決定。
- 平均値の算出、異常値の検出および誤差解析。
DNAコピー数の絶対値はcsvファイル形式で提供されます。 平均値の算出、異常値の検出および誤差解析の結果は別のファイルとして提供されます。
Counting PCR
Counting PCR (cPCR )はDNA Software社によって、DNAの絶対定量のために開発された方法です。cPCRの基本的な考え方は単一コピーのDNAからの蛍光強度を決定し、この値を濃度未知のサンプルにおける反応開始時点の蛍光値と比較することで、サンプル中のDNAコピー数を決定することです。この解析には、単一のqPCRウェルからのデータがあれば十分です。
これはバスケットの中のリンゴの数を数える場合に例えることができます。この場合、バスケット+リンゴの総重量を計測し、次にバスケットの重量を差し引き、最後に1個のリンゴの重量で割ることによって(リンゴ間の重量差が大きい場合を除き)数を算出できます。同様に、全てのDNAコピーからの蛍光からバックグラウンドを差し引いた後の値を計測し、単一コピーからの蛍光で割った値を算出することでDNAコピー数を算出できます。標準的な対象DNAの濃度(1,000,000コピー/nL以下)ではDNAからの蛍光はバックグラウンド蛍光より弱いことがわかっています。
この結果、全DNAからの蛍光も単一DNAからの蛍光も直接測定することはできません。しかしながら、qPCRを利用してコントロール可能な形で増幅した蛍光を利用し、qPCR CopyCount によって導出されたモデルを使用してqPCR曲線の変曲点を解析し、反応開始点のDNAコピー数を外挿します。
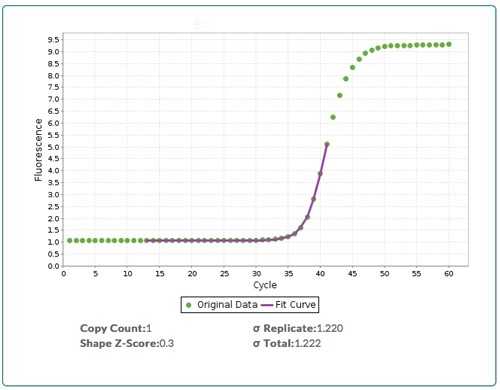
図2: qPCR CopyCountからのアウトプット
サイクル0(D0)でDNAコピー数=1である対象配列qPCRの生データ(緑)とフィット結果(紫)
最適な結果を得るために、単一コピーからの蛍光は、標準サンプルを利用することなく、アッセイ特異的なキャリブレーションを一度だけ実施することによって実験的に決定され、この結果は全ての機器およびサンプルに適用可能です。キャリブレーションにおいては、平均で1.5コピー/ウェルとなるように希釈されたサンプルを使用し、単一のプレートからの蛍光を測定することによって行われます(詳細はプレートのキャリブレーションプロトコルをご参照ください)。
384ウェルプレートを用いた場合では、絶対定量において5%以下のキャリブレーション誤差で実施可能です。プライマーデザイン、プライマー濃度およびプライマーミックスの構成に変更がなければ、キャリブレーションは以降の実験について再度実施する必要はありません。一度アッセイでキャリブレーションを実施すれば、単一ウェルからの蛍光をqPCR CopyCountで解析した結果から、標準誤差5%以内の精度でDNAコピー数の絶対定量が可能です。
遺伝子発現解析などでは比較対象の2種類の遺伝子の相対的な定量値が必要とされ、絶対定量は必要とされていません。このような場合、qPCR CopyCountはキャリブレーションなしで単一コピーからの蛍光を推定可能であり、その精度は単一ウェルからの値を用いた場合で絶対定量では20%であり、相対定量では1~5%の精度を達成します。誤差は2重に測定することでさらに低減可能です。
表1. cPCRと他の3種類のPCRを用いたDNA定量方法(デジタルPCR, 検量線を利用した絶対定量およびΔΔCt法)の特徴比較
| cPCR | デジタルPCR | 標準曲線 | ΔΔCt法 | |
| 相対定量 | ✓ | ✓ | ✓ | ✓ |
| 絶対定量 | ✓ | ✓ | ✓ | |
| 標準試料の非使用 | ✓ | ✓ | ||
| 低効率に起因した低品質データ | ✓ | ✓ | ||
| 遺伝子量正規化不要 | ✓ | ✓ | ||
| 誤差解析 | ✓ | ✓ | ||
| 特殊な機器の使用が不要 | ✓ | ✓ | ✓ | |
| 高スループット | ✓ | ✓ | ||
| レプリケート数低減 | ✓ | ✓ | ✓ | |
| 希釈系列が不要 |
cPCR は標準的なqPCRの機器をそのまま使用でき、また同一条件で測定するウェル数(リプリケート)を大幅に低減することが可能であるため、デジタルPCRと比べて高いスループットを示します。また、cPCRはデジタルPCRとは異なり希釈の必要がないため、サンプル調製が簡単です。さらに、特別なその他の器具は不要です。これらの結果、cPCRを最小限のプロトコル変更のみで既存の分析のラインナップに加えることが可能であり、同一のキャリブレーション結果を使用できるデータ群のメタ分析も可能になります。標準曲線を使用する方法やΔΔCt法とは異なり、cPCRは標準サンプルや希釈系列、遺伝子発現の標準化やPCR効率の差異の校正等は不要です。 cPCRは徹底的な誤差解析のプロセスを含んでおり、不完全なデータ取得やプライマーデザインによる信頼性の低いcPCRデータの解釈にも有用です。
表2はデジタルPCRと比較したcPCRに適した利用分野をまとめた表です。cPCRはデジタルPCRと比較してスループットが高いため、ウイルスの感染、NGS用フラグメントライブラリの定量や他の非侵襲的な診断法で利用価値を評価されています。
表2. cPCRとデジタルPCRの用途比較
| アプリケーション | cPCR | デジタルPCR |
| ハイスループットウイルス力価測定 | ✓ | |
| NGS: フラグメントライブラリの定量 | ✓ | |
| 非侵襲的診断法 | ✓ | ✓ |
| コピー数変異 | ✓ | ✓ |
| 既存データセットのメタ解析 | ✓ |
ケーススタディ:GAPDHの希釈系列
図3では単一のハウスキーピング遺伝子GAPDH (glyceraldehyde phosphate dehydrogenase)を用いた実験によって、qPCR CopyCountソフトウェアの優位性が示されています。これらの実験は各6.7 nLのPCR反応を 同時に9216 実施可能なFluidigm 社のBioMark™ HDシステムとDynamic Array™ IFC 96×9チップの組み合わせを用い、同社において実施されました。15種類の濃度の希釈系列(3倍希釈)に関して、各72のレプリケートを設定し、合計1080のqPCR反応をチップ状で実施しました。

図3: qPCR CopyCountと実験で得られたGAPDH相対的濃度のブラインドテストの結果の比較
非常にきれいな線形性(R2 = 0.99990)が濃度比で400万倍以上のレンジまで持続しています。データはDr. Gang Sun, Fluidigm Corporation提供
まとめ
Counting PCR (cPCR) はqPCRの分野における大きな不連続的な進化です。cPCRの原理は、信頼性が高い絶対定量値を算出可能な革新的なソフトウェアqPCR CopyCountに組み込まれています。qPCR CopyCount は特別な機器を必要とせず既存のqPCR装置を使用可能であり、デジタルPCRと比較してレプリケートの数をはるかに低減することが可能であるため、コストの低減に有用です。コントロールや標準サンプルを測定するためにプレートを使用する必要がなく、プレート資源はサンプルの測定に集中して使用できます。
qPCR CopyCount はまた、希釈系列を必要とせず、標準試料の調製も不要である上にハイスループットであるため、時間の節約にも有用です。さらにはFinally, qPCR CopyCount はPCR効率の補正、複雑な遺伝子の正規化、そして異なる装置、バッファーおよびプロトコルを使用したことによるPCRデータへの影響をマニュアルの計算する必要がなく、PCRのデータ解析におけるボトルネックを劇的に軽減し、作業量を減らします。qPCR CopyCount の結果を用いた絶対定量値は異なるサンプル、異なるターゲットおよび異なる施設間での結果の比較を用意に可能にします。このqPCRのメカニズムの解明におけるブレークスルーはDNAを用いたアプリケーションに大きな影響を与えると考えられます。
参考文献
[1] Patent pending. Boggy, G. J. & Woolf, P.J. (2010). “A mechanistic model of PCR for accurate quantification of quantitative PCR data.” PLoS ONE5(8): 355.
